Note
Go to the end to download the full example code
ASAI - By gram stain (binary)
Data:
date_received date_outcome patient_id laboratory_number specimen_code specimen_name specimen_description ... microorganism_name antimicrobial_code antimicrobial_name sensitivity_method sensitivity mic reported
0 2009-01-03 NaN 20091 X428501 BLDCUL NaN blood ... klebsiella AAMI amikacin NaN sensitive NaN NaN
1 2009-01-03 NaN 20091 X428501 BLDCUL NaN blood ... klebsiella AAMO amoxycillin NaN resistant NaN NaN
2 2009-01-03 NaN 20091 X428501 BLDCUL NaN blood ... klebsiella AAUG augmentin NaN sensitive NaN NaN
3 2009-01-03 NaN 20091 X428501 BLDCUL NaN blood ... klebsiella AAZT aztreonam NaN sensitive NaN NaN
4 2009-01-03 NaN 20091 X428501 BLDCUL NaN blood ... klebsiella ACAZ ceftazidime NaN sensitive NaN NaN
... ... ... ... ... ... ... ... ... ... ... ... ... ... .. ...
319117 2009-12-31 NaN 24645 H2012337 BLDCUL NaN blood ... enterococcus AAMO amoxycillin NaN sensitive NaN NaN
319118 2009-12-31 NaN 24645 H2012337 BLDCUL NaN blood ... enterococcus ALIN linezolid NaN sensitive NaN NaN
319119 2009-12-31 NaN 24645 H2012337 BLDCUL NaN blood ... enterococcus ASYN synercid NaN resistant NaN NaN
319120 2009-12-31 NaN 24645 H2012337 BLDCUL NaN blood ... enterococcus ATEI teicoplanin NaN sensitive NaN NaN
319121 2009-12-31 NaN 24645 H2012337 BLDCUL NaN blood ... enterococcus AVAN vancomycin NaN sensitive NaN NaN
[319122 rows x 15 columns]
Columns:
Index(['date_received', 'date_outcome', 'patient_id', 'laboratory_number', 'specimen_code', 'specimen_name', 'specimen_description', 'microorganism_code', 'microorganism_name', 'antimicrobial_code', 'antimicrobial_name', 'sensitivity_method', 'sensitivity', 'mic', 'reported'], dtype='object')
SARI (overall):
intermediate resistant sensitive freq sari
specimen_code microorganism_name antimicrobial_name
BFLCUL anaerobes metronidazole 0.0 0.0 1.0 1.0 0.0000
bacillus ciprofloxacin 0.0 0.0 1.0 1.0 0.0000
clindamycin 0.0 3.0 1.0 4.0 0.7500
erythromycin 0.0 1.0 3.0 4.0 0.2500
fusidic acid 0.0 3.0 1.0 4.0 0.7500
... ... ... ... ... ...
XINCUL streptococcus beta-haemolytic group b cephalexin 0.0 1.0 0.0 1.0 1.0000
clindamycin 0.0 1.0 8.0 9.0 0.1111
erythromycin 0.0 1.0 8.0 9.0 0.1111
penicillin 0.0 0.0 9.0 9.0 0.0000
tetracycline 0.0 8.0 1.0 9.0 0.8889
[4491 rows x 5 columns]
ASAI (overall):
N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
specimen_code antimicrobial_name
BFLCUL amoxycillin 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 1.0000
cefixime 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 1.0000
cefotaxime 1.0 1.0 NaN 1.0 1.0 NaN 1.0 1.0 NaN 2.0 1.0000
ceftriaxone 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 1.0000
chloramphenicol 1.0 1.0 NaN 1.0 1.0 NaN 1.0 1.0 NaN 2.0 1.0000
... ... ... .. ... ... .. ... ... .. ... ...
XINCUL tetracycline NaN 2.0 NaN NaN 2.0 NaN NaN 0.5 NaN 0.5 0.7071
timentin 4.0 NaN NaN 4.0 NaN NaN 0.0 NaN NaN 0.0 0.0000
tobramycin 4.0 NaN NaN 4.0 NaN NaN 0.5 NaN NaN 0.5 0.7071
trimethoprim NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0 1.0000
vancomycin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0 1.0000
[600 rows x 11 columns]
Cultures:
specimen_code
URICUL 116627.0
WOUCUL 94918.0
XINCUL 21427.0
SPTCUL 21113.0
BLDCUL 20333.0
ENTCUL 13110.0
T&FCUL 8150.0
MRSCUL 7865.0
VAGCUL 7425.0
EYECUL 2839.0
GUMCUL 1634.0
FAECUL 1317.0
URECUL 802.0
TISCUL 474.0
BFLCUL 450.0
SEMCUL 290.0
NEOCUL 213.0
PDFCUL 68.0
CSFCUL 32.0
RGNS 20.0
FUNSTC 14.0
TBCUL 1.0
Name: freq, dtype: float64
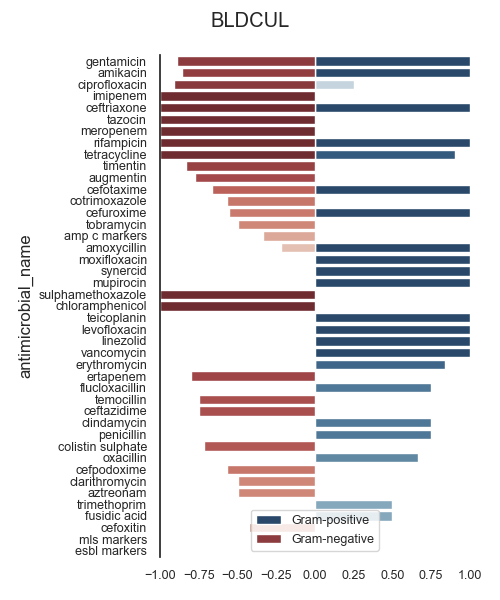
ASAI (BLDCUL)
specimen_code antimicrobial_name N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
45 BLDCUL gentamicin 9.0 3.0 1.0 9.0 4.0 1.0 0.8889 1.0000 1.0 2.8889 0.9428
23 BLDCUL amikacin 7.0 1.0 1.0 7.0 1.0 1.0 0.8571 1.0000 1.0 2.8571 0.9258
35 BLDCUL ciprofloxacin 11.0 2.0 1.0 11.0 3.0 1.0 0.9091 0.2500 1.0 2.1591 0.4767
46 BLDCUL imipenem 8.0 NaN 1.0 8.0 NaN 1.0 1.0000 NaN 1.0 2.0000 1.0000
32 BLDCUL ceftriaxone 2.0 2.0 NaN 2.0 8.0 NaN 1.0000 1.0000 NaN 2.0000 1.0000
58 BLDCUL tazocin 8.0 NaN 1.0 8.0 NaN 1.0 1.0000 NaN 1.0 2.0000 1.0000
49 BLDCUL meropenem 8.0 NaN 1.0 8.0 NaN 1.0 1.0000 NaN 1.0 2.0000 1.0000
55 BLDCUL rifampicin 1.0 2.0 NaN 1.0 3.0 NaN 1.0000 1.0000 NaN 2.0000 1.0000
61 BLDCUL tetracycline 2.0 3.0 NaN 2.0 10.0 NaN 1.0000 0.9048 NaN 1.9048 0.9512
62 BLDCUL timentin 6.0 NaN 1.0 6.0 NaN 1.0 0.8333 NaN 1.0 1.8333 0.9129
26 BLDCUL augmentin 9.0 NaN 1.0 9.0 NaN 1.0 0.7778 NaN 1.0 1.7778 0.8819
28 BLDCUL cefotaxime 9.0 2.0 1.0 9.0 8.0 1.0 0.6667 1.0000 0.0 1.6667 0.0000
39 BLDCUL cotrimoxazole 7.0 NaN 1.0 7.0 NaN 1.0 0.5714 NaN 1.0 1.5714 0.7559
33 BLDCUL cefuroxime 9.0 1.0 1.0 9.0 2.0 1.0 0.5556 1.0000 0.0 1.5556 0.0000
63 BLDCUL tobramycin 6.0 NaN 1.0 6.0 NaN 1.0 0.5000 NaN 1.0 1.5000 0.7071
25 BLDCUL amp c markers 3.0 NaN 1.0 3.0 NaN 1.0 0.3333 NaN 1.0 1.3333 0.5774
24 BLDCUL amoxycillin 9.0 3.0 1.0 9.0 9.0 1.0 0.2222 1.0000 0.0 1.2222 0.0000
51 BLDCUL moxifloxacin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
57 BLDCUL synercid NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
52 BLDCUL mupirocin NaN 1.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
56 BLDCUL sulphamethoxazole 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
34 BLDCUL chloramphenicol 2.0 NaN NaN 2.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
59 BLDCUL teicoplanin NaN 3.0 NaN NaN 10.0 NaN NaN 1.0000 NaN 1.0000 1.0000
47 BLDCUL levofloxacin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
48 BLDCUL linezolid NaN 3.0 NaN NaN 5.0 NaN NaN 1.0000 NaN 1.0000 1.0000
65 BLDCUL vancomycin NaN 4.0 NaN NaN 11.0 NaN NaN 1.0000 NaN 1.0000 1.0000
41 BLDCUL erythromycin 1.0 4.0 NaN 1.0 11.0 NaN 0.0000 0.8393 NaN 0.8393 0.0000
40 BLDCUL ertapenem 5.0 NaN NaN 5.0 NaN NaN 0.8000 NaN NaN 0.8000 0.8944
43 BLDCUL flucloxacillin NaN 2.0 NaN NaN 3.0 NaN NaN 0.7500 NaN 0.7500 0.8660
60 BLDCUL temocillin 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
31 BLDCUL ceftazidime 8.0 NaN 1.0 8.0 NaN 1.0 0.7500 NaN 0.0 0.7500 0.0000
37 BLDCUL clindamycin NaN 2.0 NaN NaN 7.0 NaN NaN 0.7500 NaN 0.7500 0.8660
54 BLDCUL penicillin 1.0 4.0 NaN 1.0 11.0 NaN 0.0000 0.7500 NaN 0.7500 0.0000
38 BLDCUL colistin sulphate 7.0 NaN 1.0 7.0 NaN 1.0 0.7143 NaN 0.0 0.7143 0.0000
53 BLDCUL oxacillin NaN 1.0 NaN NaN 3.0 NaN NaN 0.6667 NaN 0.6667 0.8165
30 BLDCUL cefpodoxime 7.0 NaN 1.0 7.0 NaN 1.0 0.5714 NaN 0.0 0.5714 0.0000
36 BLDCUL clarithromycin 2.0 NaN NaN 2.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
27 BLDCUL aztreonam 6.0 NaN NaN 6.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
64 BLDCUL trimethoprim NaN 1.0 NaN NaN 2.0 NaN NaN 0.5000 NaN 0.5000 0.7071
44 BLDCUL fusidic acid NaN 1.0 NaN NaN 2.0 NaN NaN 0.5000 NaN 0.5000 0.7071
29 BLDCUL cefoxitin 7.0 NaN 1.0 7.0 NaN 1.0 0.4286 NaN 0.0 0.4286 0.0000
50 BLDCUL mls markers NaN 2.0 NaN NaN 4.0 NaN NaN 0.0000 NaN 0.0000 0.0000
42 BLDCUL esbl markers 2.0 NaN 1.0 2.0 NaN 1.0 0.0000 NaN 0.0 0.0000 0.0000
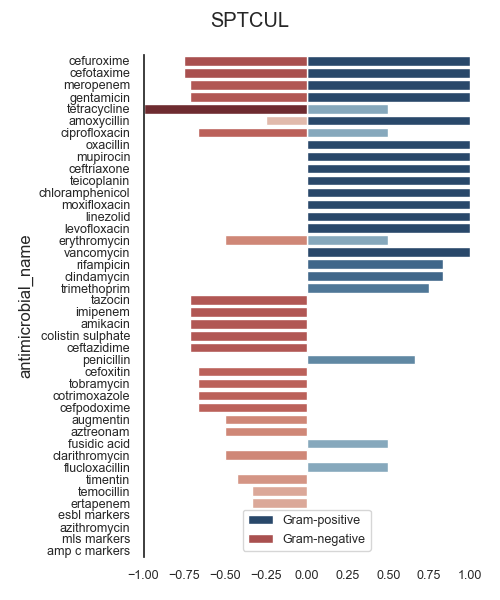
ASAI (SPTCUL)
specimen_code antimicrobial_name N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
302 SPTCUL cefuroxime 8.0 1.0 NaN 8.0 1.0 NaN 0.7500 1.0000 NaN 1.7500 0.8660
297 SPTCUL cefotaxime 8.0 1.0 NaN 8.0 1.0 NaN 0.7500 1.0000 NaN 1.7500 0.8660
318 SPTCUL meropenem 7.0 1.0 NaN 7.0 1.0 NaN 0.7143 1.0000 NaN 1.7143 0.8452
314 SPTCUL gentamicin 7.0 2.0 NaN 7.0 3.0 NaN 0.7143 1.0000 NaN 1.7143 0.8452
328 SPTCUL tetracycline 2.0 3.0 NaN 2.0 7.0 NaN 1.0000 0.5000 NaN 1.5000 0.7071
292 SPTCUL amoxycillin 8.0 1.0 NaN 8.0 1.0 NaN 0.2500 1.0000 NaN 1.2500 0.5000
304 SPTCUL ciprofloxacin 9.0 3.0 NaN 9.0 4.0 NaN 0.6667 0.5000 NaN 1.1667 0.5774
322 SPTCUL oxacillin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
321 SPTCUL mupirocin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
301 SPTCUL ceftriaxone NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
326 SPTCUL teicoplanin NaN 2.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
303 SPTCUL chloramphenicol NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
320 SPTCUL moxifloxacin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
317 SPTCUL linezolid NaN 3.0 NaN NaN 4.0 NaN NaN 1.0000 NaN 1.0000 1.0000
316 SPTCUL levofloxacin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
310 SPTCUL erythromycin 2.0 3.0 NaN 2.0 7.0 NaN 0.5000 0.5000 NaN 1.0000 0.5000
332 SPTCUL vancomycin NaN 3.0 NaN NaN 4.0 NaN NaN 1.0000 NaN 1.0000 1.0000
324 SPTCUL rifampicin NaN 3.0 NaN NaN 4.0 NaN NaN 0.8333 NaN 0.8333 0.9129
306 SPTCUL clindamycin NaN 3.0 NaN NaN 5.0 NaN NaN 0.8333 NaN 0.8333 0.9129
331 SPTCUL trimethoprim NaN 2.0 NaN NaN 3.0 NaN NaN 0.7500 NaN 0.7500 0.8660
325 SPTCUL tazocin 7.0 NaN NaN 7.0 NaN NaN 0.7143 NaN NaN 0.7143 0.8452
315 SPTCUL imipenem 7.0 NaN NaN 7.0 NaN NaN 0.7143 NaN NaN 0.7143 0.8452
291 SPTCUL amikacin 7.0 NaN NaN 7.0 NaN NaN 0.7143 NaN NaN 0.7143 0.8452
307 SPTCUL colistin sulphate 7.0 NaN NaN 7.0 NaN NaN 0.7143 NaN NaN 0.7143 0.8452
300 SPTCUL ceftazidime 7.0 NaN NaN 7.0 NaN NaN 0.7143 NaN NaN 0.7143 0.8452
323 SPTCUL penicillin NaN 3.0 NaN NaN 7.0 NaN NaN 0.6667 NaN 0.6667 0.8165
298 SPTCUL cefoxitin 6.0 NaN NaN 6.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
330 SPTCUL tobramycin 6.0 NaN NaN 6.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
308 SPTCUL cotrimoxazole 6.0 1.0 NaN 6.0 1.0 NaN 0.6667 0.0000 NaN 0.6667 0.0000
299 SPTCUL cefpodoxime 6.0 NaN NaN 6.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
294 SPTCUL augmentin 8.0 NaN NaN 8.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
296 SPTCUL aztreonam 4.0 NaN NaN 4.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
313 SPTCUL fusidic acid NaN 2.0 NaN NaN 3.0 NaN NaN 0.5000 NaN 0.5000 0.7071
305 SPTCUL clarithromycin 2.0 NaN NaN 2.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
312 SPTCUL flucloxacillin NaN 1.0 NaN NaN 2.0 NaN NaN 0.5000 NaN 0.5000 0.7071
329 SPTCUL timentin 7.0 NaN NaN 7.0 NaN NaN 0.4286 NaN NaN 0.4286 0.6547
327 SPTCUL temocillin 3.0 NaN NaN 3.0 NaN NaN 0.3333 NaN NaN 0.3333 0.5774
309 SPTCUL ertapenem 3.0 NaN NaN 3.0 NaN NaN 0.3333 NaN NaN 0.3333 0.5774
311 SPTCUL esbl markers 1.0 NaN NaN 1.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
295 SPTCUL azithromycin 1.0 1.0 NaN 1.0 1.0 NaN 0.0000 0.0000 NaN 0.0000 0.0000
319 SPTCUL mls markers NaN 2.0 NaN NaN 2.0 NaN NaN 0.0000 NaN 0.0000 0.0000
293 SPTCUL amp c markers 1.0 NaN NaN 1.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
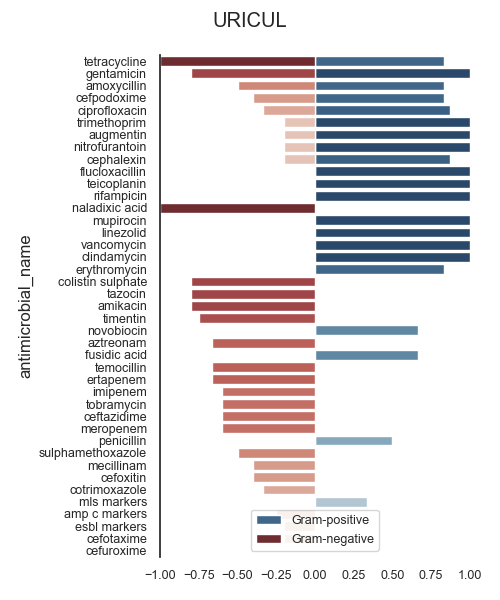
ASAI (URICUL)
specimen_code antimicrobial_name N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
478 URICUL tetracycline 1.0 2.0 NaN 1.0 6.0 NaN 1.0000 0.8333 NaN 1.8333 0.9129
462 URICUL gentamicin 5.0 1.0 NaN 5.0 3.0 NaN 0.8000 1.0000 NaN 1.8000 0.8944
443 URICUL amoxycillin 2.0 2.0 NaN 2.0 7.0 NaN 0.5000 0.8333 NaN 1.3333 0.6455
449 URICUL cefpodoxime 5.0 2.0 NaN 5.0 5.0 NaN 0.4000 0.8333 NaN 1.2333 0.5774
453 URICUL ciprofloxacin 6.0 2.0 NaN 6.0 7.0 NaN 0.3333 0.8750 NaN 1.2083 0.5401
481 URICUL trimethoprim 5.0 2.0 NaN 5.0 7.0 NaN 0.2000 1.0000 NaN 1.2000 0.4472
445 URICUL augmentin 5.0 2.0 NaN 5.0 7.0 NaN 0.2000 1.0000 NaN 1.2000 0.4472
470 URICUL nitrofurantoin 5.0 2.0 NaN 5.0 7.0 NaN 0.2000 1.0000 NaN 1.2000 0.4472
452 URICUL cephalexin 5.0 2.0 NaN 5.0 7.0 NaN 0.2000 0.8750 NaN 1.0750 0.4183
460 URICUL flucloxacillin 1.0 1.0 NaN 1.0 3.0 NaN 0.0000 1.0000 NaN 1.0000 0.0000
476 URICUL teicoplanin NaN 2.0 NaN NaN 7.0 NaN NaN 1.0000 NaN 1.0000 1.0000
473 URICUL rifampicin NaN 1.0 NaN NaN 3.0 NaN NaN 1.0000 NaN 1.0000 1.0000
469 URICUL naladixic acid 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
468 URICUL mupirocin NaN 1.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
464 URICUL linezolid NaN 1.0 NaN NaN 3.0 NaN NaN 1.0000 NaN 1.0000 1.0000
482 URICUL vancomycin NaN 2.0 NaN NaN 7.0 NaN NaN 1.0000 NaN 1.0000 1.0000
454 URICUL clindamycin NaN 2.0 NaN NaN 4.0 NaN NaN 1.0000 NaN 1.0000 1.0000
458 URICUL erythromycin NaN 2.0 NaN NaN 6.0 NaN NaN 0.8333 NaN 0.8333 0.9129
455 URICUL colistin sulphate 5.0 NaN NaN 5.0 NaN NaN 0.8000 NaN NaN 0.8000 0.8944
475 URICUL tazocin 5.0 NaN NaN 5.0 NaN NaN 0.8000 NaN NaN 0.8000 0.8944
442 URICUL amikacin 5.0 NaN NaN 5.0 NaN NaN 0.8000 NaN NaN 0.8000 0.8944
479 URICUL timentin 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
471 URICUL novobiocin 1.0 1.0 NaN 1.0 3.0 NaN 0.0000 0.6667 NaN 0.6667 0.0000
446 URICUL aztreonam 3.0 NaN NaN 3.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
461 URICUL fusidic acid NaN 1.0 NaN NaN 3.0 NaN NaN 0.6667 NaN 0.6667 0.8165
477 URICUL temocillin 3.0 NaN NaN 3.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
457 URICUL ertapenem 3.0 NaN NaN 3.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
463 URICUL imipenem 5.0 NaN NaN 5.0 NaN NaN 0.6000 NaN NaN 0.6000 0.7746
480 URICUL tobramycin 5.0 NaN NaN 5.0 NaN NaN 0.6000 NaN NaN 0.6000 0.7746
450 URICUL ceftazidime 5.0 NaN NaN 5.0 NaN NaN 0.6000 NaN NaN 0.6000 0.7746
466 URICUL meropenem 5.0 NaN NaN 5.0 NaN NaN 0.6000 NaN NaN 0.6000 0.7746
472 URICUL penicillin 1.0 2.0 NaN 1.0 6.0 NaN 0.0000 0.5000 NaN 0.5000 0.0000
474 URICUL sulphamethoxazole 2.0 NaN NaN 2.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
465 URICUL mecillinam 5.0 NaN NaN 5.0 NaN NaN 0.4000 NaN NaN 0.4000 0.6325
448 URICUL cefoxitin 5.0 NaN NaN 5.0 NaN NaN 0.4000 NaN NaN 0.4000 0.6325
456 URICUL cotrimoxazole 3.0 NaN NaN 3.0 NaN NaN 0.3333 NaN NaN 0.3333 0.5774
467 URICUL mls markers NaN 1.0 NaN NaN 3.0 NaN NaN 0.3333 NaN 0.3333 0.5774
444 URICUL amp c markers 4.0 NaN NaN 4.0 NaN NaN 0.2500 NaN NaN 0.2500 0.5000
459 URICUL esbl markers 5.0 NaN NaN 5.0 NaN NaN 0.2000 NaN NaN 0.2000 0.4472
447 URICUL cefotaxime 5.0 NaN NaN 5.0 NaN NaN 0.2000 NaN NaN 0.2000 0.4472
451 URICUL cefuroxime 5.0 NaN NaN 5.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
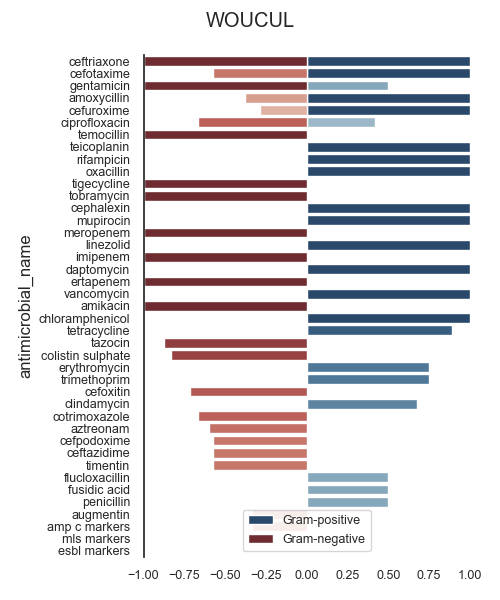
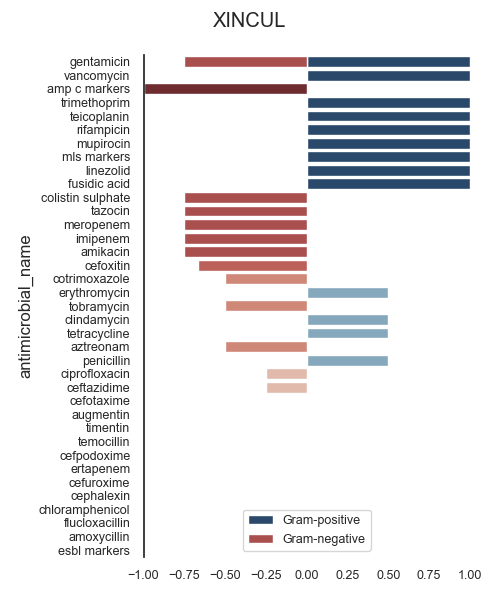
ASAI (WOUCUL)
specimen_code antimicrobial_name N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
531 WOUCUL ceftriaxone 1.0 1.0 NaN 1.0 1.0 NaN 1.0000 1.0000 NaN 2.0000 1.0000
527 WOUCUL cefotaxime 7.0 1.0 NaN 7.0 2.0 NaN 0.5714 1.0000 NaN 1.5714 0.7559
545 WOUCUL gentamicin 8.0 1.0 NaN 8.0 2.0 NaN 1.0000 0.5000 NaN 1.5000 0.7071
523 WOUCUL amoxycillin 8.0 1.0 NaN 8.0 3.0 NaN 0.3750 1.0000 NaN 1.3750 0.6124
532 WOUCUL cefuroxime 7.0 1.0 NaN 7.0 1.0 NaN 0.2857 1.0000 NaN 1.2857 0.5345
535 WOUCUL ciprofloxacin 9.0 2.0 NaN 10.0 5.0 NaN 0.6667 0.4167 NaN 1.0833 0.5270
556 WOUCUL temocillin 3.0 NaN NaN 3.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
555 WOUCUL teicoplanin NaN 2.0 NaN NaN 8.0 NaN NaN 1.0000 NaN 1.0000 1.0000
553 WOUCUL rifampicin NaN 2.0 NaN NaN 4.0 NaN NaN 1.0000 NaN 1.0000 1.0000
551 WOUCUL oxacillin NaN 1.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
558 WOUCUL tigecycline 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
560 WOUCUL tobramycin 5.0 NaN NaN 5.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
533 WOUCUL cephalexin NaN 1.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
550 WOUCUL mupirocin NaN 1.0 NaN NaN 2.0 NaN NaN 1.0000 NaN 1.0000 1.0000
548 WOUCUL meropenem 8.0 NaN NaN 8.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
547 WOUCUL linezolid NaN 2.0 NaN NaN 4.0 NaN NaN 1.0000 NaN 1.0000 1.0000
546 WOUCUL imipenem 8.0 NaN NaN 8.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
539 WOUCUL daptomycin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN 1.0000 1.0000
540 WOUCUL ertapenem 3.0 NaN NaN 3.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
562 WOUCUL vancomycin NaN 2.0 NaN NaN 8.0 NaN NaN 1.0000 NaN 1.0000 1.0000
522 WOUCUL amikacin 6.0 NaN NaN 6.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
534 WOUCUL chloramphenicol NaN 2.0 NaN NaN 5.0 NaN NaN 1.0000 NaN 1.0000 1.0000
557 WOUCUL tetracycline 1.0 2.0 NaN 2.0 11.0 NaN 0.0000 0.8889 NaN 0.8889 0.0000
554 WOUCUL tazocin 8.0 NaN NaN 8.0 NaN NaN 0.8750 NaN NaN 0.8750 0.9354
537 WOUCUL colistin sulphate 6.0 NaN NaN 6.0 NaN NaN 0.8333 NaN NaN 0.8333 0.9129
541 WOUCUL erythromycin NaN 2.0 NaN NaN 11.0 NaN NaN 0.7500 NaN 0.7500 0.8660
561 WOUCUL trimethoprim NaN 2.0 NaN NaN 4.0 NaN NaN 0.7500 NaN 0.7500 0.8660
528 WOUCUL cefoxitin 7.0 1.0 NaN 7.0 1.0 NaN 0.7143 0.0000 NaN 0.7143 0.0000
536 WOUCUL clindamycin NaN 2.0 NaN NaN 9.0 NaN NaN 0.6786 NaN 0.6786 0.8238
538 WOUCUL cotrimoxazole 6.0 NaN NaN 6.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
526 WOUCUL aztreonam 5.0 NaN NaN 5.0 NaN NaN 0.6000 NaN NaN 0.6000 0.7746
529 WOUCUL cefpodoxime 7.0 NaN NaN 7.0 NaN NaN 0.5714 NaN NaN 0.5714 0.7559
530 WOUCUL ceftazidime 7.0 NaN NaN 7.0 NaN NaN 0.5714 NaN NaN 0.5714 0.7559
559 WOUCUL timentin 7.0 NaN NaN 7.0 NaN NaN 0.5714 NaN NaN 0.5714 0.7559
543 WOUCUL flucloxacillin NaN 1.0 NaN NaN 2.0 NaN NaN 0.5000 NaN 0.5000 0.7071
544 WOUCUL fusidic acid NaN 1.0 NaN NaN 2.0 NaN NaN 0.5000 NaN 0.5000 0.7071
552 WOUCUL penicillin NaN 2.0 NaN NaN 11.0 NaN NaN 0.5000 NaN 0.5000 0.7071
525 WOUCUL augmentin 6.0 NaN NaN 6.0 NaN NaN 0.3333 NaN NaN 0.3333 0.5774
524 WOUCUL amp c markers 3.0 NaN NaN 3.0 NaN NaN 0.3333 NaN NaN 0.3333 0.5774
549 WOUCUL mls markers NaN 2.0 NaN NaN 4.0 NaN NaN 0.0000 NaN 0.0000 0.0000
542 WOUCUL esbl markers 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
ASAI (XINCUL)
specimen_code antimicrobial_name N_GENUS N_SPECIE ASAI_SCORE width gmean
gram_stain n p u n p u n p u
584 XINCUL gentamicin 4.0 1.0 NaN 4.0 1.0 NaN 0.7500 1.0 NaN 1.7500 0.8660
599 XINCUL vancomycin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
565 XINCUL amp c markers 1.0 NaN NaN 1.0 NaN NaN 1.0000 NaN NaN 1.0000 1.0000
598 XINCUL trimethoprim NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
593 XINCUL teicoplanin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
591 XINCUL rifampicin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
589 XINCUL mupirocin NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
588 XINCUL mls markers NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
586 XINCUL linezolid NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
583 XINCUL fusidic acid NaN 1.0 NaN NaN 1.0 NaN NaN 1.0 NaN 1.0000 1.0000
577 XINCUL colistin sulphate 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
592 XINCUL tazocin 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
587 XINCUL meropenem 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
585 XINCUL imipenem 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
563 XINCUL amikacin 4.0 NaN NaN 4.0 NaN NaN 0.7500 NaN NaN 0.7500 0.8660
569 XINCUL cefoxitin 3.0 NaN NaN 3.0 NaN NaN 0.6667 NaN NaN 0.6667 0.8165
578 XINCUL cotrimoxazole 4.0 NaN NaN 4.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
580 XINCUL erythromycin NaN 2.0 NaN NaN 2.0 NaN NaN 0.5 NaN 0.5000 0.7071
597 XINCUL tobramycin 4.0 NaN NaN 4.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
576 XINCUL clindamycin NaN 2.0 NaN NaN 2.0 NaN NaN 0.5 NaN 0.5000 0.7071
595 XINCUL tetracycline NaN 2.0 NaN NaN 2.0 NaN NaN 0.5 NaN 0.5000 0.7071
567 XINCUL aztreonam 2.0 NaN NaN 2.0 NaN NaN 0.5000 NaN NaN 0.5000 0.7071
590 XINCUL penicillin NaN 2.0 NaN NaN 2.0 NaN NaN 0.5 NaN 0.5000 0.7071
575 XINCUL ciprofloxacin 4.0 1.0 NaN 4.0 1.0 NaN 0.2500 0.0 NaN 0.2500 0.0000
571 XINCUL ceftazidime 4.0 NaN NaN 4.0 NaN NaN 0.2500 NaN NaN 0.2500 0.5000
568 XINCUL cefotaxime 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
566 XINCUL augmentin 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
596 XINCUL timentin 4.0 NaN NaN 4.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
594 XINCUL temocillin 1.0 NaN NaN 1.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
570 XINCUL cefpodoxime 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
579 XINCUL ertapenem 1.0 NaN NaN 1.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
572 XINCUL cefuroxime 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
573 XINCUL cephalexin NaN 1.0 NaN NaN 1.0 NaN NaN 0.0 NaN 0.0000 0.0000
574 XINCUL chloramphenicol NaN 1.0 NaN NaN 1.0 NaN NaN 0.0 NaN 0.0000 0.0000
582 XINCUL flucloxacillin NaN 1.0 NaN NaN 1.0 NaN NaN 0.0 NaN 0.0000 0.0000
564 XINCUL amoxycillin 3.0 NaN NaN 3.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
581 XINCUL esbl markers 1.0 NaN NaN 1.0 NaN NaN 0.0000 NaN NaN 0.0000 0.0000
"\n# ----------\n# Example\n# ----------\n# This example shows a diverging figure using exclusively the gram-positive\n# and gram-negative categories. Note that the gram negative categorie has\n# values in the range [-1,0] while the gram-positive category has values\n# within the range [0, 1]\n# Create figure\nf, axes = plt.subplots(1, 2, figsize=(7, 8), sharey=True)\n\n# Plot with pallete according to value\nsns.barplot(x=y_p, y=x, ax=axes[0], orient='h', palette=colormap_p,\n saturation=0.5, label='Gram-positive')\nsns.barplot(x=-y_n, y=x, ax=axes[0], orient='h', palette=colormap_n,\n saturation=0.5, label='Gram-negative')\n\n# Plot with plain palette (values are already size of bars)\nsns.barplot(x=y_p, y=x, ax=axes[1], orient='h', color='b',\n saturation=0.5, label='Gram-positive')\nsns.barplot(x=-y_n, y=x, ax=axes[1], orient='h', color='r',\n saturation=0.5, label='Gram-negative')\n\n# Configure\nsns.despine(bottom=True)\n\n# Configure axes\naxes[0].set(title='ASAI (width)')\naxes[1].set(title='ASAI (width)')\n\n# Show legend.\nplt.legend()\n\n# Adjust\nplt.tight_layout()\n\n# Show\nplt.show()\n"
7 # Import libraries
8 import sys
9 import numpy as np
10 import pandas as pd
11 import seaborn as sns
12 import matplotlib as mpl
13 import matplotlib.pyplot as plt
14
15 # Import specific libraries
16 from pyamr.core.asai import ASAI
17 from pyamr.core.sari import SARI
18 from pyamr.core.freq import Frequency
19 from pyamr.datasets.load import make_susceptibility
20
21 # -------------------------
22 # Configuration
23 # -------------------------
24 # Configure seaborn style (context=talk)
25 sns.set(style="white")
26
27 # Set matplotlib
28 mpl.rcParams['xtick.labelsize'] = 9
29 mpl.rcParams['ytick.labelsize'] = 9
30 mpl.rcParams['axes.titlesize'] = 11
31 mpl.rcParams['legend.fontsize'] = 9
32
33 # Pandas configuration
34 pd.set_option('display.max_colwidth', 40)
35 pd.set_option('display.width', 300)
36 pd.set_option('display.precision', 4)
37
38 # Numpy configuration
39 np.set_printoptions(precision=2)
40
41 # -------------------------------------------
42 # Load data
43 # -------------------------------------------
44 # Load data
45 data = make_susceptibility()
46
47 # Show
48 print("\nData:")
49 print(data)
50 print("\nColumns:")
51 print(data.columns)
52
53 # -------------------------------------------
54 # Compute SARI
55 # -------------------------------------------
56 # Libraries
57 from pyamr.core.sari import SARI
58
59 # Create sari instance
60 sari = SARI(groupby=['specimen_code',
61 'microorganism_name',
62 'antimicrobial_name',
63 'sensitivity'])
64
65 # Compute SARI overall
66 sari_overall = sari.compute(data,
67 return_frequencies=True)
68
69 # Show
70 print("SARI (overall):")
71 print(sari_overall)
72
73
74
75 # ------------------------------
76 # Include gram stain
77 # ------------------------------
78 # Libraries
79 from pyamr.datasets.registries import MicroorganismRegistry
80
81 # Load registry
82 mreg = MicroorganismRegistry()
83
84 # Format sari dataframe
85 dataframe = sari_overall.copy(deep=True)
86 dataframe = dataframe.reset_index()
87
88 # Create genus and species
89 dataframe[['genus', 'species']] = \
90 dataframe.microorganism_name \
91 .str.capitalize() \
92 .str.split(expand=True, n=1)
93
94 # Combine with registry information
95 dataframe = mreg.combine(dataframe, on='microorganism_name')
96
97 # Fill missing gram stain
98 dataframe.gram_stain = dataframe.gram_stain.fillna('u')
99
100
101 # -------------------------------------------
102 # Compute ASAI
103 # -------------------------------------------
104 # Import specific libraries
105 from pyamr.core.asai import ASAI
106
107 # Create asai instance
108 asai = ASAI(column_genus='genus',
109 column_specie='species',
110 column_resistance='sari',
111 column_frequency='freq')
112
113 # Compute
114 scores = asai.compute(dataframe,
115 groupby=['specimen_code',
116 'antimicrobial_name',
117 'gram_stain'],
118 weights='uniform',
119 threshold=0.5,
120 min_freq=0)
121
122 # Stack
123 scores = scores.unstack()
124
125 # .. note: In order to sort the scores we need to compute metrics
126 # that combine the different subcategories (e.g. gram-negative
127 # and gram-positive). Two possible options are: (i) use the
128 # gmean or (ii) the width.
129 # Measures
130 scores['width'] = np.abs(scores['ASAI_SCORE'].sum(axis=1))
131 scores['gmean'] = np.sqrt(scores['ASAI_SCORE'].product(axis=1))
132
133 # Show
134 print("\nASAI (overall):")
135 print(scores)
136
137
138 # ------------------------
139 # Methods
140 # ------------------------
141 def scalar_colormap(values, cmap, vmin, vmax):
142 """This method creates a colormap based on values.
143
144 Parameters
145 ----------
146 values : array-like
147 The values to create the corresponding colors
148
149 cmap : str
150 The colormap
151
152 vmin, vmax : float
153 The minimum and maximum possible values
154
155 Returns
156 -------
157 scalar colormap
158 """
159 # Create scalar mappable
160 norm = mpl.colors.Normalize(vmin=vmin, vmax=vmax, clip=True)
161 mapper = mpl.cm.ScalarMappable(norm=norm, cmap=cmap)
162 # Gete color map
163 colormap = sns.color_palette([mapper.to_rgba(i) for i in values])
164 # Return
165 return colormap
166
167
168 # -------------------------------------------
169 # Plot
170 # -------------------------------------------
171 # Reset
172 scores = scores.reset_index()
173
174 # Count records per specimen
175 specimen_count = sari_overall \
176 .groupby('specimen_code').freq.sum() \
177 .sort_values(ascending=False)
178
179 # Show
180 print("\nCultures:")
181 print(specimen_count)
182
183 # Filter
184 scores = scores[scores.specimen_code\
185 .isin(specimen_count.index.values[:5])]
186
187 # Loop
188 for specimen, df in scores.groupby(by='specimen_code'):
189
190 # Sort
191 df = df.sort_values(by='width', ascending=False)
192
193 # Show
194 print("\n\nASAI (%s)" % specimen)
195 print(df)
196
197 # Variables to plot.
198 x = df.antimicrobial_name
199 y_n = df['ASAI_SCORE']['n'].values
200 y_p = df['ASAI_SCORE']['p'].values
201
202 # Constants
203 colormap_p = scalar_colormap(y_p, cmap='Blues', vmin=-0.1, vmax=1.1)
204 colormap_n = scalar_colormap(y_n, cmap='Reds', vmin=-0.1, vmax=1.1)
205
206 # ----------
207 # Example
208 # ----------
209 # This example shows an stacked figure using more than two categories.
210 # For instance, it uses gram-positive, gram-negative and gram-unknown.
211 # All the indexes go within the range [0,1].
212 # Create figure
213 f, ax = plt.subplots(1, 1, figsize=(5, 6))
214
215 # Plot
216 sns.barplot(x=y_p, y=x, palette=colormap_p, ax=ax, orient='h',
217 saturation=0.5, label='Gram-positive')
218 sns.barplot(x=-y_n, y=x, palette=colormap_n, ax=ax, orient='h',
219 saturation=0.5, label='Gram-negative')
220
221 # Configure
222 sns.despine(bottom=True)
223
224 # Format figure
225 plt.subplots_adjust(wspace=0.0, hspace=0.0)
226
227 # Set x-axis
228 ax.set_xlim([-1, 1])
229
230 # Adjust
231 plt.legend(loc=8)
232 plt.suptitle(specimen)
233 plt.tight_layout()
234
235 # Show
236 plt.show()
237
238
239
240
241
242
243
244
245 """
246 # ----------
247 # Example
248 # ----------
249 # This example shows a diverging figure using exclusively the gram-positive
250 # and gram-negative categories. Note that the gram negative categorie has
251 # values in the range [-1,0] while the gram-positive category has values
252 # within the range [0, 1]
253 # Create figure
254 f, axes = plt.subplots(1, 2, figsize=(7, 8), sharey=True)
255
256 # Plot with pallete according to value
257 sns.barplot(x=y_p, y=x, ax=axes[0], orient='h', palette=colormap_p,
258 saturation=0.5, label='Gram-positive')
259 sns.barplot(x=-y_n, y=x, ax=axes[0], orient='h', palette=colormap_n,
260 saturation=0.5, label='Gram-negative')
261
262 # Plot with plain palette (values are already size of bars)
263 sns.barplot(x=y_p, y=x, ax=axes[1], orient='h', color='b',
264 saturation=0.5, label='Gram-positive')
265 sns.barplot(x=-y_n, y=x, ax=axes[1], orient='h', color='r',
266 saturation=0.5, label='Gram-negative')
267
268 # Configure
269 sns.despine(bottom=True)
270
271 # Configure axes
272 axes[0].set(title='ASAI (width)')
273 axes[1].set(title='ASAI (width)')
274
275 # Show legend.
276 plt.legend()
277
278 # Adjust
279 plt.tight_layout()
280
281 # Show
282 plt.show()
283 """
Total running time of the script: ( 0 minutes 5.412 seconds)